Multiplex Ligation-Dependent Probe Amplification (MLPA) และ MassARRAY
MLPA (Multiplex Ligation-dependent Probe Amplification) และ MassARRAY (เทคนิคที่ใช้ MALDI-TOF mass spectrometry ในการตรวจวิเคราะห์พันธุกรรม) เป็นเทคนิคทางอณูชีววิทยาที่ใช้สำหรับการวิเคราะห์ทางพันธุกรรม แต่มีวัตถุประสงค์และวิธีการที่แตกต่างกัน ดังนี้
MLPA
MLPA (Multiplex Ligation-dependent Probe Amplification) เป็นเทคนิคที่ใช้ในการวิเคราะห์ความแตกต่างของจำนวนสำเนา (Copy Number Variation, CNV) ของ DNA โดยเฉพาะการตรวจหาการกลายพันธุ์หรือการลบ/เพิ่มของยีนที่เกี่ยวข้องกับโรคทางพันธุกรรม
ภาพ 1 การแสดงผลของเทคนิค MLPA เราสามารถสังเกตอิเล็กโตรเฟโรแกรม (Electropherogram)
ที่ได้จากการวิเคราะห์ MLPA ซึ่งแสดงถึงการขาดหายไปของ Exon ที่ 46 (สีแดง)
ภาพโดย https://commons.wikimedia.org/wiki/File:MLPA_in_GeneMarker.jpg)
กระบวนการทำงานของ MLPA
MLPA มีขั้นตอนหลัก ๆ ดังนี้:
- การออกแบบโพรบ (Probe Design)
• ใช้โพรบคู่ (Probe pair) ที่มีลำดับเฉพาะสำหรับเป้าหมายของ DNA
• แต่ละโพรบประกอบด้วยสองส่วนที่สามารถจับกับ DNA ติดกัน
• โพรบแต่ละตัวมีส่วนซ้าย (Left probe oligonucleotide; LPO) และส่วนขวา (Right probe oligonucleotide; RPO) โดยมีลำดับสำหรับการจับกับ DNA เป้าหมาย และลำดับที่ใช้เป็นไพรเมอร์สำหรับ PCR ทั้งนี้ RPO ยังมีลำดับเสริม (stuffer sequence) เพื่อทำให้แต่ละโพรบมีความยาวเฉพาะตัว - การแยกสาย DNA (Denaturation)
• ตัวอย่าง DNA ถูกทำให้แยกสายด้วยความร้อน (~95°C) เพื่อเปิดโอกาสให้โพรบสามารถจับกับสาย DNA ได้อย่างจำเพาะ - การจับของโพรบ (Hybridization)
• โพรบคู่แต่ละตัวจะจับกับสาย DNA เป้าหมายที่ตำแหน่งเฉพาะ
• หากตำแหน่งเป้าหมายมีการลบ (deletion) โพรบจะไม่สามารถจับได้ - การเชื่อมต่อโพรบ (Ligation)
• เมื่อโพรบทั้งสองส่วนจับกับ DNA ได้อย่างสมบูรณ์ จะมีเอนไซม์ DNA ligase ทำหน้าที่เชื่อมต่อให้เป็นชิ้นเดียว
• หากมีการกลายพันธุ์ที่ตำแหน่งจับ อาจส่งผลให้การเชื่อมต่อล้มเหลว - การขยายชิ้นส่วน DNA (PCR Amplification)
• ใช้ PCR เพื่อขยายเฉพาะโพรบที่ถูกเชื่อมต่อแล้ว
• โพรบแต่ละตัวมีความยาวต่างกัน ทำให้สามารถแยกแยะได้ในการตรวจวิเคราะห์ - การแยกขนาดของผลิตภัณฑ์ PCR (Capillary Electrophoresis)
• นำผลิตภัณฑ์ PCR ไปวิเคราะห์ด้วย capillary electrophoresis
• ขนาดของแต่ละแบนด์ที่ได้สะท้อนถึงจำนวนสำเนาของ DNA เป้าหมาย - การวิเคราะห์ข้อมูล
• เปรียบเทียบปริมาณสัญญาณของผลิตภัณฑ์ PCR กับตัวอย่างอ้างอิง
• หากมีการลบ (deletion) หรือการเพิ่ม (duplication) จะเห็นความแตกต่างของสัดส่วนสัญญาณ
การนำไปใช้ของ MLPA
• ตรวจหาความผิดปกติทางพันธุกรรม เช่น Duchenne Muscular Dystrophy (DMD), Spinal Muscular Atrophy (SMA)
• วิเคราะห์การแปรผันของยีนที่เกี่ยวข้องกับมะเร็ง เช่น BRCA1 และ BRCA2
• ใช้ในการวินิจฉัยโรคที่เกี่ยวข้องกับ CNV ได้อย่างรวดเร็วและแม่นยำ แม้ในตัวอย่างที่มี DNA ปริมาณน้อย
การตรวจหาการกลายพันธุ์หรือการลบ/เพิ่มของยีนที่เกี่ยวข้องกับโรคทางพันธุกรรม
MassARRAY
MassARRAY เป็นเทคโนโลยีการวิเคราะห์พันธุกรรมที่อาศัยหลักการของ mass spectrometry (การวิเคราะห์มวลสาร) โดยมีความแม่นยำสูงในการตรวจจับการกลายพันธุ์ของยีนแบบเฉพาะตำแหน่ง เช่น SNP (Single Nucleotide Polymorphism), การกลายพันธุ์แบบจุด (Point mutation) และการแปรผันอื่น ๆ ที่เกี่ยวข้องกับโรคทางพันธุกรรมหรือการตอบสนองต่อยา
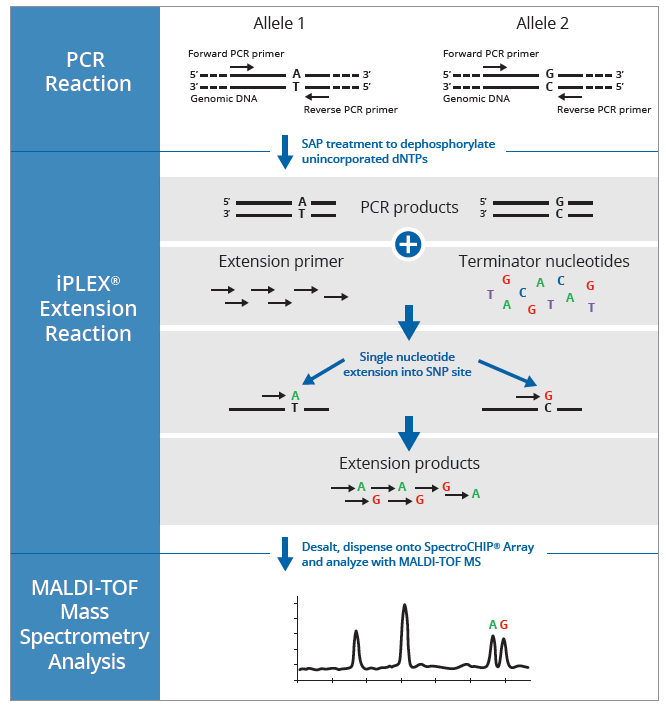
ภาพ 2 การแสดกระบวนการทำงานของ MassARRAY ในการตรวจสอบ SNP
โดยเริ่มจากการทำ PCR, การขยายตำแหน่ง SNP ด้วย iPLEX Extension Reaction
และการวิเคราะห์ผลิตภัณฑ์ด้วย MALDI-TOF Mass Spectrometry
กระบวนการทำงานของ MassARRAY
MassARRAY มีขั้นตอนหลัก ๆ ดังนี้:
-
การออกแบบไพรเมอร์ (Primer Design)
• ออกแบบไพรเมอร์เฉพาะสำหรับบริเวณของ DNA ที่ต้องการตรวจ
• ไพรเมอร์ถูกออกแบบให้เหมาะสมกับการวิเคราะห์ด้วย mass spectrometry โดยมีความแม่นยำสูง
• การออกแบบต้องพิจารณาเรื่องมวลของผลิตภัณฑ์ที่ได้จากการขยายตัวของ DNA -
การขยายชิ้นส่วน DNA (PCR Amplification)
• ใช้ PCR ขยายเฉพาะบริเวณของ DNA ที่สนใจ
• ผลิตภัณฑ์ PCR จะใช้เป็นแม่แบบในขั้นตอนถัดไป
• ขั้นตอนนี้ช่วยเพิ่มปริมาณ DNA เป้าหมายให้เพียงพอต่อการวิเคราะห์ -
การกำจัดดีเอ็นเอส่วนเกิน (SAP Treatment)
• ใช้เอนไซม์ Shrimp Alkaline Phosphatase (SAP) กำจัด dNTP ที่เหลืออยู่จาก PCR
• เพื่อป้องกันการรบกวนในขั้นตอนถัดไปของการขยายปฏิกิริยา -
การขยายสัญญาณแบบ Single Base Extension (SBE)
• เติมนิวคลีโอไทด์เพียงตัวเดียวต่อปลายไพรเมอร์โดยอาศัยเอนไซม์
• การเติมนิวคลีโอไทด์นี้จะขึ้นอยู่กับเบสที่แท้จริงในตำแหน่งที่สนใจ
• แต่ละนิวคลีโอไทด์มีมวลเฉพาะ ทำให้สามารถแยกได้ด้วย mass spectrometry -
การวิเคราะห์ด้วย MALDI-TOF (Matrix-Assisted Laser Desorption/Ionization Time-of-Flight)
• นำผลิตภัณฑ์จาก SBE มาวิเคราะห์ด้วย MALDI-TOF
• เครื่องจะตรวจวัดมวลของผลิตภัณฑ์อย่างแม่นยำ
• ผลที่ได้จะแสดงเป็นพีคตามมวลของนิวคลีโอไทด์ที่เติมเข้าไป -
การวิเคราะห์ข้อมูล
• ข้อมูลจาก mass spectrometry ถูกวิเคราะห์ด้วยซอฟต์แวร์เฉพาะ
• สามารถระบุการกลายพันธุ์, SNP หรือการแปรผันอื่น ๆ ได้อย่างชัดเจน
• ให้ข้อมูลทั้งเชิงคุณภาพและเชิงปริมาณ
การนำไปใช้ของ MassARRAY
• การวิเคราะห์ SNP เพื่อศึกษาความสัมพันธ์กับโรค เช่น มะเร็ง, เบาหวาน, ความดันโลหิตสูง
• การวินิจฉัยโรคทางพันธุกรรมเฉพาะตำแหน่ง เช่น CFTR, β-thalassemia
• การทดสอบเภสัชพันธุศาสตร์ (Pharmacogenomics) เพื่อพยากรณ์การตอบสนองต่อยา
• การศึกษา population genetics และการตรวจสอบแหล่งกำเนิดทางชีวภาพ
MassARRAY เป็นเครื่องมือที่ทรงพลัง ให้ข้อมูลที่ละเอียดและแม่นยำ เหมาะกับการตรวจวิเคราะห์ในระดับสูงและการใช้ในงานวิจัยหรืองานวินิจฉัยทางคลินิก
ตารางเปรียบเทียบ MLPA กับ MassARRAY
|
คุณสมบัติ |
MLPA |
MassARRAY |
|
การใช้งานหลัก |
ตรวจ CNV, การลบ/เพิ่มของยีน, เมทิลเลชัน |
ตรวจ SNPs, การกลายพันธุ์, เมทิลเลชัน |
|
วิธีการวิเคราะห์ |
Ligation + PCR + Capillary electrophoresis |
MALDI-TOF mass spectrometry |
|
ชนิดของการเปลี่ยนแปลงที่ตรวจพบ |
CNVs, การเปลี่ยนแปลงเมทิลเลชัน |
SNPs, การกลายพันธุ์ขนาดเล็ก, เมทิลเลชัน |
|
จำนวนตำแหน่งที่ตรวจได้ |
~60 ตำแหน่งต่อรอบ |
หลายร้อย SNPs ต่อรอบ |
|
ความเร็ว |
ปานกลาง (~1 วัน) |
เร็ว (ภายในไม่กี่ชั่วโมง) |
เลือกใช้เทคนิคใดดี?
- หากต้องการ ตรวจการเปลี่ยนแปลงจำนวนสำเนาของยีน (CNVs) หรือการลบ/การเพิ่มจำนวนของยีน → MLPA เหมาะสมกว่า
- หากต้องการ ตรวจ SNPs, การกลายพันธุ์แบบจุดเดียว หรือการวิเคราะห์เมทิลเลชัน → MassARRAY เป็นตัวเลือกที่ดีกว่า

